Supposons que vous disposiez de données chronologiques à lignes et colonnes que vous souhaitez alimenter en un calque dans Keras. Avant de transmettre cela au RNN, vous devez reformater les données précédentes en un tenseur 3D. Donc, cela devient un .N700SimpleRNN(200, return_sequence=True)N×700×1
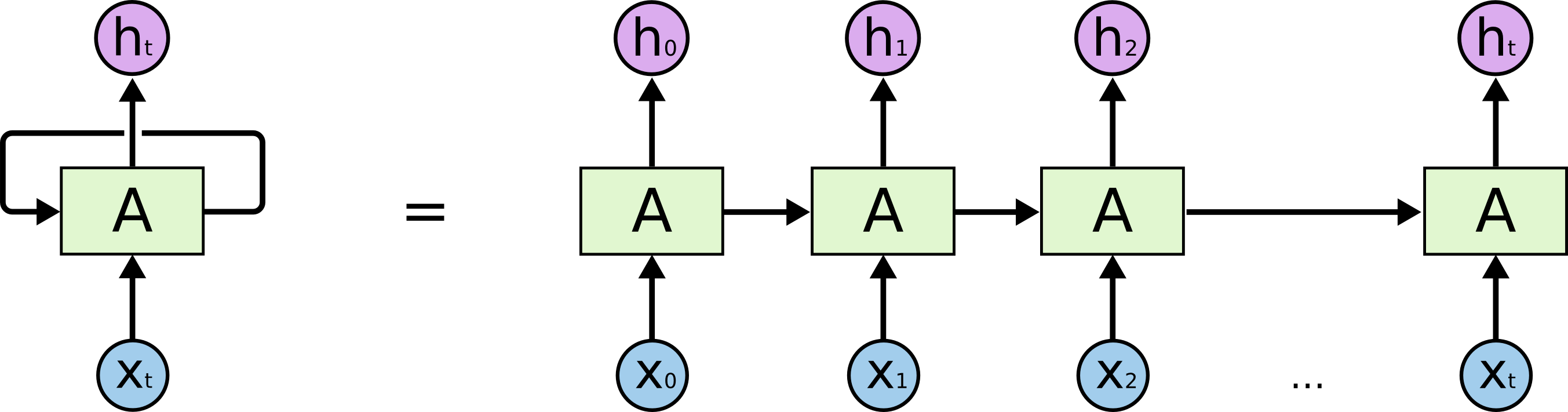
L'image est tirée de https://colah.github.io/posts/2015-08-Understanding-LSTMs
Dans RNN, vos colonnes (les "700 colonnes") sont les pas de temps de RNN. Vos données sont traitées de . Après avoir transmis les données au RNN, il dispose maintenant de 700 sorties, à et non à . N'oubliez pas que la forme de vos données est maintenant ce qui correspond à des échantillons (les lignes) x timesteps (les colonnes) x canaux .t=1 to 700h1h700h1h200N×700×200
Et ensuite, lorsque vous appliquez a TimeDistributedDense, vous appliquez un Densecalque à chaque pas de temps, ce qui signifie que vous appliquez un Densecalque à chaque , , ..., respectivement. Ce qui signifie que vous appliquez l’opération entièrement connectée sur chacun de ses canaux (le "200"), respectivement, de à . Le 1er " " jusqu'au 700ème " ".h1h2hth1h7001×1×2001×1×200
Pourquoi fait-on ça? Parce que vous ne voulez pas aplatir la sortie RNN.
Pourquoi ne pas aplatir la sortie RNN? Parce que vous voulez que chaque valeur de pas de temps soit séparée.
Pourquoi garder chaque valeur de pas distinct? Car:
- vous voulez seulement interagir les valeurs entre son propre pas de temps
- vous ne voulez pas avoir une interaction aléatoire entre différents timesteps et canaux.
